1.はじめに
先日 DeepMind は、アミノ酸配列からタンパク質の立体構造を推定する AlphaFold2 を公開し、その高い精度が構造生物学界を始め、生命科学研究者やAI研究者にも大きな驚きを与えています。今回は、このホットな話題を見て行こうと思います。
2.タンパク質の立体構造推定について
私たちの体は水を除けば半分くらいはタンパク質で出来ています。なので、タンパク質の解析が進むと生体内の化学反応が解明できます。そして、アルツハイマー型認知症やパーキンソン病といったタンパク質由来の病気の解明が進み、新たな治療薬開発などにも役立ちます。
タンパク質は20種類あるアミノ酸が繋がったヒモ状のもので、そのヒモが立体的に折りたたまれています。そして、タンパク質の機能は特定のアミノ酸同士が近くにあるかによって決まります。そのため、タンパク質を解析するには、アミノ酸配列を調べるだけではなく、立体構造も調べる必要があります。
ところが、このタンパク質の立体構造を調べるのは大変な作業で、既にアミノ酸配列が分かっているタンパク質が数十億種類あるのに対して、立体構造まで明らかになっているものは約10万種類に留まってるのが現実です。
なぜ、タンパク質の立体構造を調べるのが大変かというと、労力と時間がかかるからです。その方法は、まず長期間(何ヶ月レベル)掛けて高純度の大きな(0.1mm程度)結晶を作ります、次にその結晶にX線を照射し膨大な数の回折点を測定します、そしてその結果を元にコンピュータで構造計算をさせるというものです。
Deepmind は、既に明らかになっているタンパク質の立体構造のデータベースを元に、ディープラーニングを使って新たなタンパク質の立体構造を推定する学習モデルを構築しました。この方法の出現によって、今後タンパク質の解析スピードが大幅に加速することが期待できます。
早速コードを動かしてみましょう。AlphaFold2の正式版は 2.2TBのファイル容量が必要なため、今回は有志が作った(素晴らしい!)簡略化した Google Colab 版を動かしてみます。
3.AlphaFold2(Google Colab版)
AlphaFold2 の Google Colab版は、このリンクで動かすことが出来ます。各ブロックはコードが非表示になっているので、コードを見たい場合はブロックをクリックして下さい。それでは、順番に各ブロックを見て行きましょう。
Input protein sequence, then hit Runtime -> Run all
最初のブロックです。query_sequence にアミノ酸配列を FASTA と呼ばれるフォーマットで設定します。ここでは、下記のヒト・リゾチームというタンパク質のアミノ酸配列をコピペして設定します。
|
1 |
MKALIVLGLVLLSVTVQGKVFERCELARTL KRLGMDGYRGISLANWMCLAKWESGYNTRA TNYNAGDRSTDYGIFQINSRYWCNDGKTPG AVNACHLSCSALLQDNIADAVACAKRVVRD PQGIRAWVAWRNRCQNRDVRQYVQGCGV |
NCBIで様々なタンパク質のアミノ酸配列(FASTA)を見付けることが出来ますので、色々試してみて下さい。
num_models には、1度に予測を行うモデル数(1〜5)を設定します。2〜5を設定すると、推定精度を比較して一番良いモデルを選択できます。ここでは、初期設定の5をそのまま使います。
必要な設定を行ったら、Google Colab の上段にある「ランタイム/全てのセルを実行」をクリックします。そうすると、以下の全てのブロックが自動的に実行されます。簡単!
Install dependencies
ライブラリーのインストール、学習済みモデルのダウンロード等を行います。
Import libraries
ライブラリーのインポート、関数の定義を行います。
Call MMseqs2 to get MSA/templates
インターネットを通じて、既に分かっているタンパク質の構造をまとめたMSAデータベースにアクセスし、今回推定するタンパク質に類似する構造データを取得します。

Gather input features, predict structure
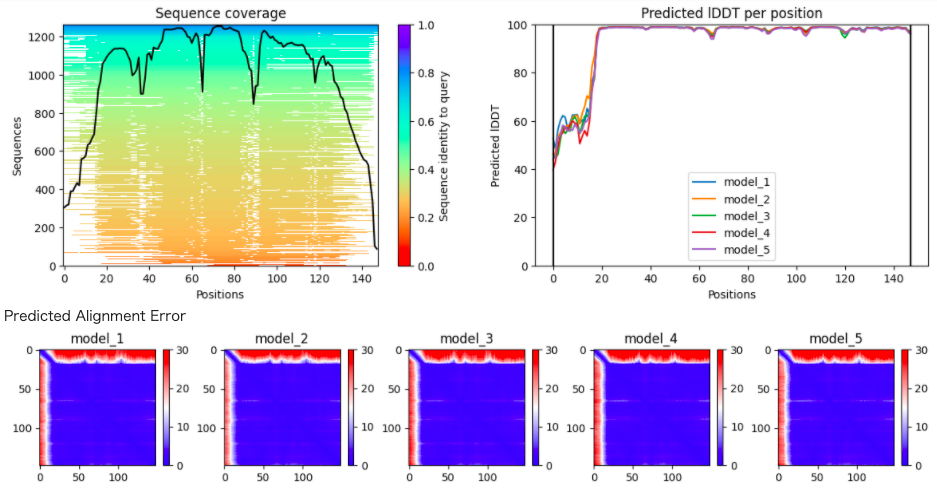
5つのモデルで構造予測を行い、各モデルの精度を IDDT(局所距離差検定)という指標で数値化します。ここでは、model_1が1番精度が高いことが分かります。

Make Plot
5つのモデルのIDDTの詳細結果を表示します。タンパク質のヒモの位置によって精度が変化することが分かります。これを平均したものが、先ほどの数値です。
Display 3D structure
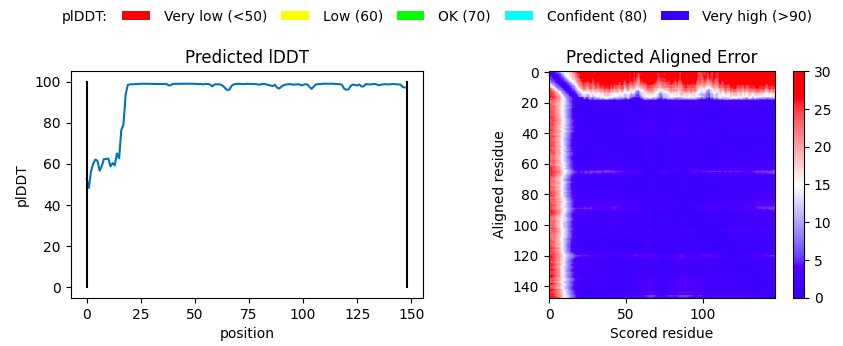
5つのモデルの中から推定精度の1番高いものを3D表示します。Google Colab の画面上でマウス操作すると、自由な角度から見ることが出来ます(拡大・縮小も可能です)。色は推定精度を表していて、90%超が青、以下水色・緑・黄の順で確率が低くなり、赤は50%未満です。

Package and download results
最後のブロックでは、推定した5つのタンパク質の3Dモデルを pdb ファイルでまとめてダウンロードします。pdb ファイルは、Jmol というビュアーで見ることが出来ますので、興味のある方は試してみて下さい。
*ブラウザによって、自動ダウンロードしない場合もあるようです。その場合はGoogle Chrome でやってみて下さい。
今回の背景には、CASP(Critical Assessment of Structure Prediction)というタンパク質の構造推定の精度を競うコンテストがあり、2020年に Deepmind は驚異的なハイスコアで1位を獲得したことがあります。素晴らしいコンテストですよね。
では、また。
(オリジナルgithub)https://github.com/deepmind/alphafold














コメントを残す